
2025年8月27日,南方医科大学周晓红和中国中医科学院陈同课题组联合在iMetaOmics在线发表了题为“GISDD: a comprehensive global integrated sequence and genotyping database platform for dengue virus, facilitating a stratified coordinated surveillance strategy”的文章。研发并提供了一个集成化在线登革病毒(DENV)数据库和基因分型研究平台GISDD(Global Integrated Sequence and Genotyping Database for Dengue Virus),整合了深度学习基因分型识别GISDDrlearn等多种在线工具与资源,基于全球统一分型框架,可实现DENV基因型、亚基因型及基因分支的高效甄别与溯源预警,为构建全球协同监测分层体系提供支持。
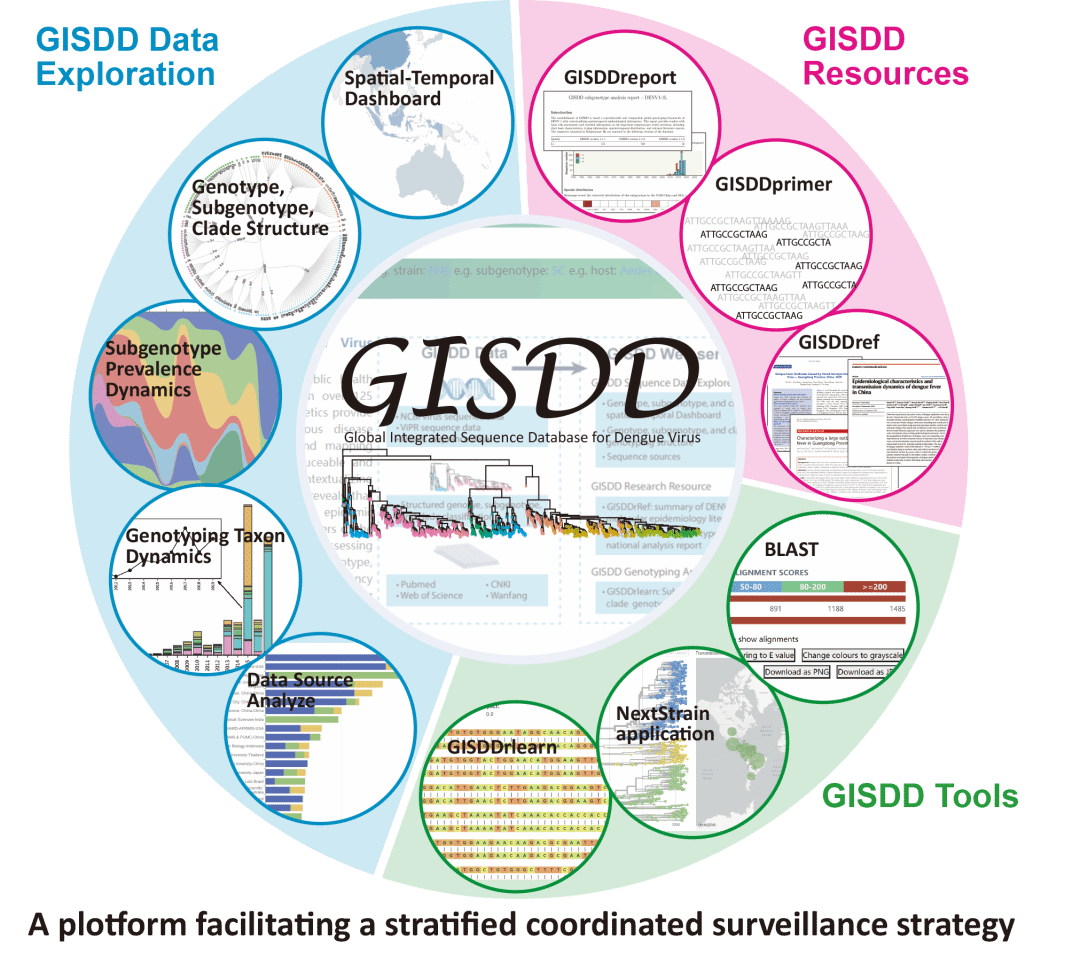
●GISDD集成了针对DENV的深度学习基因分型识别GISDDrlearn等一系列在线分析工具,基于全球统一分型框架,可实现DENV基因型、亚基因型及基因分支的高效快速甄别与溯源;
● GISDD中SIR工作流支持用户通过网页界面直接上传DENV序列数据,系统将自动完成分析并生成该序列的基因分型判定、溯源与风险预警及其在全球的时空流行特征结果报告;
● GISDD为构建全球DENV分层监测协同战略与体系提供支持。
摘 要
重要蚊媒传染病登革热是由登革病毒(DENV)引起,近年来登革热的持续快速蔓延及其所引发的日趋严重的疾病负担,给全球公共卫生体系带来前所未有的挑战,凸显了构建全球DENV监测控制协同战略与体系的必要性和紧迫性。本研究基于前期已建立的DENV全球统一分型框架的基础,系统性构建了利于分层监测协同的DENV全球整合基因序列数据库和基因分型研究平台GISDD。GISDD整合了针对DENV所建立的深度学习基因分型识别GISDDrlearn等一系列在线分析工具与资源,可实现DENV基因型、亚基因型及基因分支的高效快速甄别与溯源,有利于深入解析DENV的分子流行病学特征及其时空传播规律。该数据库可通过 http://www.bic.ac.cn/GISDD/访问,可成为研究人员、公共卫生机构及政策制定者的重要资源。GISDD为构建全球DENV分层监测协同战略与体系奠定了基础,对遏制DENV在全球范围内的快速传播具有重要意义。
全文解读
引 言
登革病毒(DENV)隶属黄病毒属,引发登革热,主要通过埃及伊蚊和白纹伊蚊传播,其在全球快速播散至125个国家,全球半数以上人口面临DENV感染风险,严重威胁民众生命健康,使全球公共卫生体系面临严峻挑战。登革热疾病负担的加重可归因于多种因素,包括城市化进程加快、气候变化、国际贸易和交通一体化。这些因素与媒介蚊虫的快速扩散共同推动了登革热不可逆转的流行趋势。近年来,病毒测序技术和系统发育分析的不断进步,已成为解决病毒性传染病流行病学中关键问题的重要工具。在前期研究中,我们构建了融合时空流行病学信息的DENV全球统一基因分型框架,辨识了登革热快速传播的三大关键驱动因素:传统的本土流行区的持续性输出、新发流行区的渐进性蔓延以及隐匿性疫情的规模化存在。进一步基于该分型框架,以系统动态发育研究阐释了中国广东DENV-1跨区域流行传播规律。
基因测序数据的指数级增长以及复杂的多源数据架构和需求,推动了病毒遗传信息数据库的快速建立。然而,研究人员在访问GenBank和ViPR等公共数据库时,常面临获取特定信息(如地理来源、宿主物种信息及旅行史)的困难。此外,区分与疫情无关数据(如实验室传代毒株或人工改造毒株)也存在较大挑战。从文献和报告中提取“细粒度”数据的潜力尚未被充分挖掘。近年来多个专注于病毒学的专业数据库相继建立,如GISAID、CoV-lineages、Pathogenwatch和Nextstrain,这些平台显著提升了病毒的监测与基因分型能力。但DENV的全球统一基因分型技术平台仍亟待构建。因此,基于前期已建立的DENV全球统一基因分型框架,本研究构建了DENV全球整合基因序列数据库和基因分型研究平台(GISDD),网址为https://www.bic.ac.cn/GISDD/,该数据库是一个经过系统整理和严格筛选的DENV序列信息资源库与基因分型技术平台,旨在为构建全球DENV分层监测协同战略与体系奠定基础。
结 果
GISDD数据库特征
GISDD(1.3.2版)包含41,336条序列,其中16,058条为DENV-1,13,923条为DENV-2,7579条为DENV-3,3776条为DENV-4。所收集的序列数量在时间分布上呈现随时间逐渐积累增多趋势,自2005年起每年新增大约一千个序列,于2013年至2019年间呈峰值。近年来,序列数量有所下降,这可能归因于新冠病毒(SARS-CoV-2)疫情的影响以及因研究周期、数据共享与发表存在的潜在延迟。数据库序列的长度集中在三个主要范围:200-500 bp、1300-1500 bp和10400-10800 bp,分别对应于常见DENV靶基因区域,如非结构1(NS1)基因片段(约240 bp)、全长包膜(E)基因(约1,485 bp)和完整基因组(约11kb)。DENV毒株的地理分布范围广泛,但显著集中于东亚和东南亚,而非洲相对较少。566家涵盖大学、科研院所及医疗机构的单位,683篇研究论文,贡献了DENV基因序列。
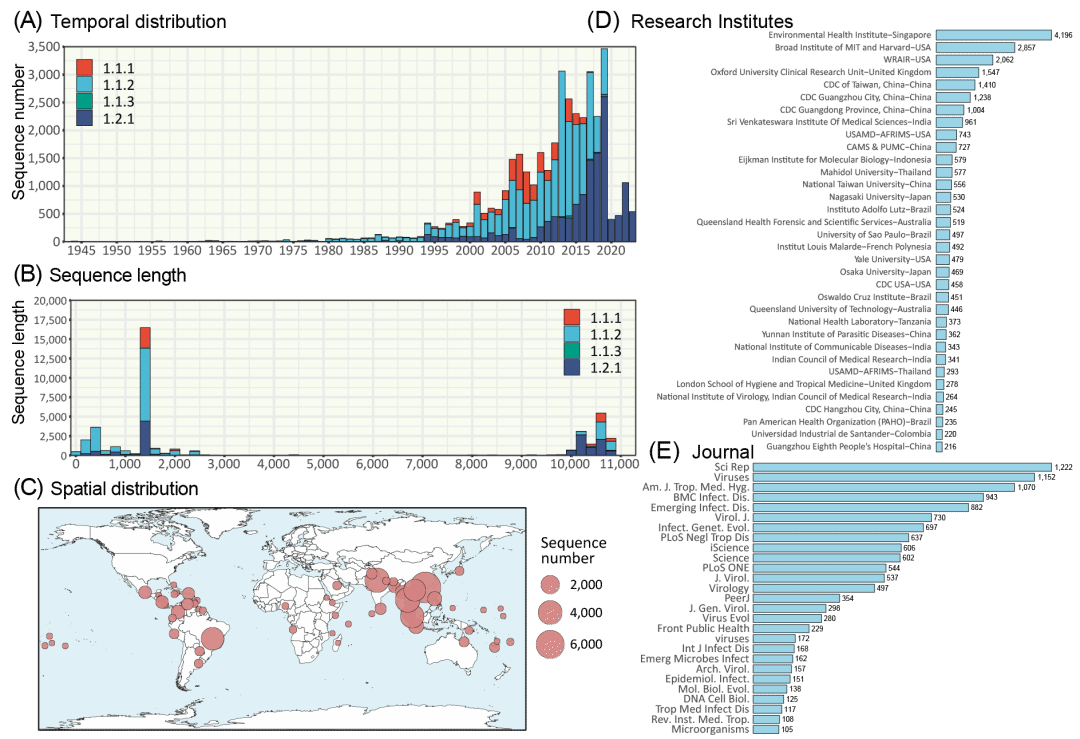
图 1. GISDD数据库的关键特征
GSIDD中DENV毒株发表的时间分布(A)、序列长度分布(B)和国家/地区空间分布(C)。根据报告的DENV序列数量,列出了排名前30的机构(D)和排名前30的期刊(E)。
GISDD网站交互界面设计
GISDD设计了一个用户友好的网络界面,便利于用户在DENV基因型、基因亚型和基因分支三级基因分型框架内高效地查询、浏览、审查和探索数据。提供了直观的导航栏,引导用户通过“浏览”、“探索”、“资源”、“工具”、“工作流程”和“关于”等导航按钮访问关键栏目。
使用GISDD数据探索
为了提升用户查询和浏览数据的便捷性,GISDD主页配置了一个多功能搜索框,支持用户通过多种类型的关键字进行精准检索。用户也可以点击选项按钮,轻松查看全面信息并下载所有可用数据。数据探索界面中,例如“Spatial-Temporal Dashboard”,用户能够对DENV序列的时空分布进行全面分析。通过有选择地筛选特定基因型、亚基因型或基因分支,或定义精确的时间范围,用户可以生成DENV靶序列的详细时空分析,为理解DENV时空动态提供有价值的见解。
GISDD在线资源
“GISDDreport”包含两个系列的分析报告,重点聚焦于DENV的基因亚型/基因分支和地域分布分析。第一系列表述了基因亚型/基因分支层面的分析,提供DENV靶序列的风险评估及相关基因分支的关键信息,包括其分布特征、基因分型结果、地理分布模式以及相关支持文献。第二系列则专注于地域层面的分析,提供各国登革热流行地区的重要基因亚型信息及其风险评估情况。
为全面呈现DENV分子流行病学研究前沿,系统检索并整合了591篇相关研究,纳入描述性分析,并可通过GISDDref资源界面,配备交互式仪表板,可对全球及中国DENV分子流行病学研究进行系统性总结与可视化展示。
此外,GISDDprimer旨在便捷检索已发表及新设计的针对DENV基因片段的引物对。该工具的总结表格提供了详尽信息,包括引物名称、引物对/组名称、引物序列、其在DENV基因组中的位置、引物长度、目标血清型、目标片段、对所有已收录DENV序列可能产生的脱靶率以及相关参考文献。
GISDD在线分析工具
GISDD“Tools”页面提供了一套在线分析工具,包括GISDDrlearn、BLAST和基本系统发育分析。随着病毒基因组数据的海量增长,研发能够处理更大规模基因组数据的新型方法变得至关重要。随机森林作为一种重要的机器学习模型,由多个决策树组成,每个决策树基于训练样本的不同子集构建。这种结构与进化树之间存在相似的结构关系。基于此理念研发了GISDDrlearn,一个R脚本应用程序,用于通过随机森林算法将DENV序列进行血清型、基因型、基因亚型和基因分支的分型。一方面,以完整的DENV E基因所构建的全球统一分型框架为基础,针对E基因序列,利用GISDDrlearn可以快速自动进行DENV的三级基因分型。另一方面,GISDDrlearn实现的高分辨率基因分型,可进一步将传统的基于E基因的基因分型方法与新兴的基因组分型技术与流行病学研究相结合。
GISDD序列分型-综合研判-风险评估(SIR)工作流
SARS-CoV-2序列的系统发育分析与流行病学数据的整合,为公共卫生官员和政策制定者制定有效干预措施与防控决策,提供了理解跨境的时空传播信息和关键科学依据。然而,DENV的相关研究仍面临以下两个方面的挑战。一方面,虽然高通量测序技术(NGS)在高收入国家已经广泛使用多年,但在中低收入国家的应用情况却参差不齐。实施NGS的障碍包括对外部资金的依赖、供应链的复杂性、训练有素的人力资源短缺以及有限的质量保证机制。另一方面,DENV研究中缺乏一个全球统一的数据共享的协同战略与体系,阻碍了实时数据同步。上述挑战常常使得通过系统发育分析来实现DENV的监测与溯源变得困难。另外,由于测序序列与其相关序列之间存在显著的时间间隔,这可能导致部分病毒种群的明显缺失,因而出现难以根据常规的进化分析准确追踪其起源。本研究团队基于DENV全球统一分型框架,辨识了在中国流行的7个重要的DENV-1主基因支系(Clades of Concern, COCs)5C1、1K1、1E1、1H4、1J7、1L1和1L2, 基于COCs系统发育树进一步指定了97个DENV-1的流行传播簇,结合相关流行病学信息,发现7个COCs分别指定的簇中有18.8%至100%无法推断其来源。
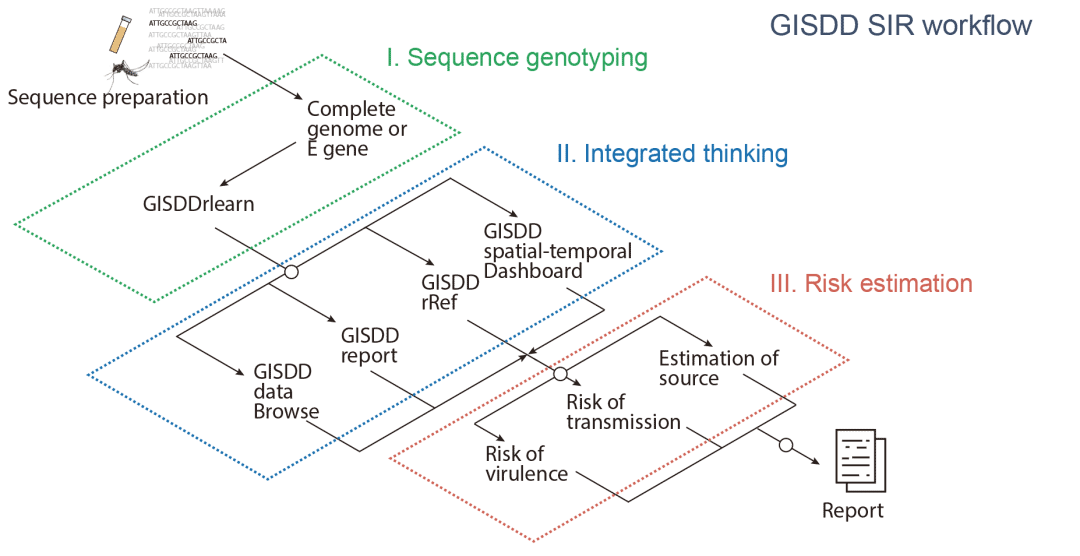
图2. GISDD序列基因分型-综合研判-风险评估(SIR)工作流
SIR工作流整合了三个核心分析阶段:(1)基因分型,(2)综合分析与判断,(3)风险评估。该工作流程依托GISDD平台,提供用户友好的网页界面,支持在线提交序列数据及自动生成与输出分析结果。
本团队的前期研究揭示DENV-1种群分布呈现显著地理限制性特征,表征为洲际-基因型(Continent-Genotype)、区域-基因亚型(Region-Subgenotype)和国家-分支(Nation-Clade)的分层流行配对(stratified spatial-genetic epidemic pairs)规律。进一步对中国广东DENV-1跨区域流行传播规律的研究中发现,在中国流行的DENV-1基因支系以及7个COCs中所指定的流行传播簇的序列数量均遵循幂律分布,其中超过80%的序列集中在7个COCs。
基于对DENV传播模式的既往综合分析及本研究结果,我们在GISDD研究平台中提出了SIR工作流。该流程分为三个阶段:序列基因分型、综合研判和风险评估。第一阶段:用户可方便选择上传多种序列类型,包括使用NGS原始数据和组装序列等。上传包含完整的E基因序列数据后,便可通过SIR工作流第一阶段采用GISDDrlearn等工具进行靶序列的基因型确定。第二阶段:当完成SIR第一阶段的DENV序列的基因型判定后,进一步构建分子进化树,将系统发育分析整合时空分布及来自GISDDreport的流行病学信息,对靶序列的相关基因亚型/基因支系进行综合研判。第三阶段,对靶序列特定基因亚型/基因分支的综合资源信息对其进行流行传播的风险评估。最后,SIR工作流自动完成分析后生成该序列的基因分型判定、溯源与风险预警及其在全球的时空流行特征结果报告,用户在通过网页直接提交其序列后,可便利接收到相应的分析报告。
应用案例1:中国流行DENV-2群体遗传结构分析
应用GISDD平台,对近年来DENV-2在中国的流行情况进行了系统分析,发现其基因型III、IV、V和VI的22个亚基因型,在中国流行,其中亚基因型3AM、3D和5B的毒株比例超过了10%。从时间动态的角度来看,观察到近年来DENV-2在中国流行呈上升趋势,毒株数量递增,其中亚基因型3AM呈现显著的增长趋势。由此可推断,自2019年亚基因型3AM在中国的流行程度呈现加强趋势。
应用案例2:深圳2024年一起本地登革热疫情的SIR分析
对2024年发生在深圳宝安的一个建筑工地的本地登革热疫情进行了案例研究。通过SIR工作流分析了此次疫情中获得的E基因序列,确定流行病毒株为DENV-2的3AM亚型。全球范围内,该亚型近年来已成为DENV-2的主流行株,在当年全球报告的DENV-2序列中约占80%。在中国,该亚型在2015、2017和2019均有大量序列报告。溯源分析,该基因亚型传入很可能源自马来西亚、新加坡和印度尼西亚等东南亚国家,柬埔寨等湄公河沿岸国家亦存在可能性。系统发育分析表明,此次疫情是由独立输入事件引发的本地传播。而关于亚型3AM的传播机制和致病性,目前的研究信息仍然有限,值得进一步加强研究。
方 法
数据库构建、数据收集与处理
GISDD数据库定义七个数据类别具体包括:数据质量、菌株分离、序列信息、基因分型、流行病学信息、投稿期刊和机构以及备注。这些类别涵盖了四十四项详细内容,例如分离宿主、测序技术、采集日期、报告机构等。该数据库与 ViPR、国家微生物数据中心和NCBI等公开科学数据库进行了整合。数据库不仅收录了相关文章和作者的信息,还确保了数据的一致性和完整性。严格的筛选流程从标题和摘要的初步筛选开始,经过基于设定的纳入和排除标准的全文评估,最终确定符合条件的研究。基因分型信息,包括基因型、基因亚型和基因分支归属,是在已确立的DENV全球统一的基因分型框架下进行分类的。这些序列根据其长度、基因完整性和其他相关因素进行分类和标记。
网站框架
GISDD平台前端开发使用了 JavaScript和HTML,所使用的核心JavaScript库包括用于主框架的Vue.js(https://vuejs.org)、用于交互式图表的echarts(https://echarts.apache.org)、plotly.js(https://plotly.com/)和D3.js(https://d3js.org/)。后端数据传输使用了高级网络框架Django(https://www.djangoproject.com)。Mysql开源数据管理系统被用于保存和访问表数据。已经在包括谷歌浏览器、火狐浏览器和Internet Explorer在内的各种网络浏览器上进行了广泛的浏览器兼容性测试。
GISDDprimer数据库
GISDD引物库提供了已发表的用于PCR、qPCR和Cas检测的引物对/组的集合,包含详细的序列信息。我们使用了关键词组合,如“Dengue”或“DENV”以及(“primer”或“detection”),并结合中文同义词“登革热”或“登革病毒”以及(“引物”或“检测”),在PubMed(https://www.ncbi.nlm.nih.gov/pubmed/)、中国知网数据库(CNKI,http://www.cnki.net)和万方数据知识服务平台(http://g.wanfangdata.com.cn/)等中英文数据库中进行了全面搜索。为了验证扩增区域并评估潜在的脱靶效应,我们基于一系列评估指标为收集到的引物及新提交的引物设计了一套严格的评估流程。这些指标包括引物在DENV基因组中的位置、引物长度、靶向血清型、片段大小特异性及潜在脱靶率。
GISDDref数据库
在中英文数据库中采用了与GISDDprimer所述一致的检索策略进行文献检索。检索关键词包括“dengue”或“DENV”,以及对应的中文词汇“登革热”或“登革病毒”。文献综述遵循严格的选择标准,具体步骤包括:由两位作者独立进行文献检索,通过专家咨询解决分歧,并根据标题、摘要和全文内容进行多轮筛选以排除不相关的研究。此外,还纳入了通过所选报告的参考文献或专家建议确定的相关记录,确保所有符合资格的DENV流行病学或分子流行病学研究,包括病例报告、病例系列、综述、横断面研究和队列研究。
GISDDrlearn基因分型工具
为了提高DENV基因分型的准确性,GISDDrlearn采用随机森林分类算法预测DENV的基因型、基因亚型和基因分支。该模型使用R语言及其“caret”包(版本6.094)和“randomForest”包构建,并通过准确率值选择最优模型参数(mtry和splitrule)。通过构建混淆矩阵,对训练集中每个序列的血清型、基因型及基因亚型预测结果进行了系统评估,以验证 GISDDrlearn 模型的性能。
DOI: GISDD: A comprehensive global integrated sequence and genotyping database platform for dengue virus, facilitating a stratified coordinated surveillance strategy - Guo - 2025 - iMetaOmics - Wiley Online Library